Pracownia Transkryptomiki Przestrzennej Spatial Transcriptomics Laboratory
Zakład Histologii i Embriologii Department of Histology and Embryology
Uniwersytet Medyczny im. Karola Marcinkowskiego w Poznaniu Poznan University of Medical Sciences

Zakład Histologii i Embriologii Department of Histology and Embryology
Uniwersytet Medyczny im. Karola Marcinkowskiego w Poznaniu Poznan University of Medical Sciences

Transkryptomika przestrzenna to metoda badająca ekspresję całego transkryptomu (wszystkich genów) w preparacie histologicznym, z zachowaniem informacji o ich dokładnej lokalizacji w strukturze tkankowej. Pozwala to na nałożenie mapy aktywności genów bezpośrednio na obraz histologiczny preparatu wraz z analizą ich potencjalnych interakcji. Spatial transcriptomics is a method that examines the expression of the entire transcriptome (all genes) in a histological slide while preserving information about their precise location in tissue structure. This allows overlaying gene activity maps directly onto histological images of the slide along with analysis of their potential interactions.
Dane transkryptomiczne o rozdzielczości pojedynczej komórki bez utraty informacji przestrzennej Transcriptomic data at single-cell resolution without loss of spatial information
Zachowanie kontekstu anatomicznego poprzez nałożenie danych molekularnych na barwienie H&E Preservation of anatomical context through overlay of molecular data on H&E staining
Mapowanie interakcji międzykomórkowych w natywnym mikrośrodowisku tkankowym Mapping of intercellular interactions in native tissue microenvironment
Identyfikacja rzadkich populacji komórkowych w ich naturalnych niszach Identification of rare cell populations in their natural niches
Transkryptomika przestrzenna łączy zalety histologii/histopatologii klasycznej (morfologia, lokalizacja) z możliwościami transkryptomiki funkcjonalnej (profilowanie ekspresji tysięcy genów jednocześnie), otwierając nowe perspektywy dla badań podstawowych i translacyjnych. Spatial transcriptomics combines the advantages of classical histology/histopathology (morphology, localization) with the capabilities of functional transcriptomics (profiling expression of thousands of genes simultaneously), opening new perspectives for basic and translational research.
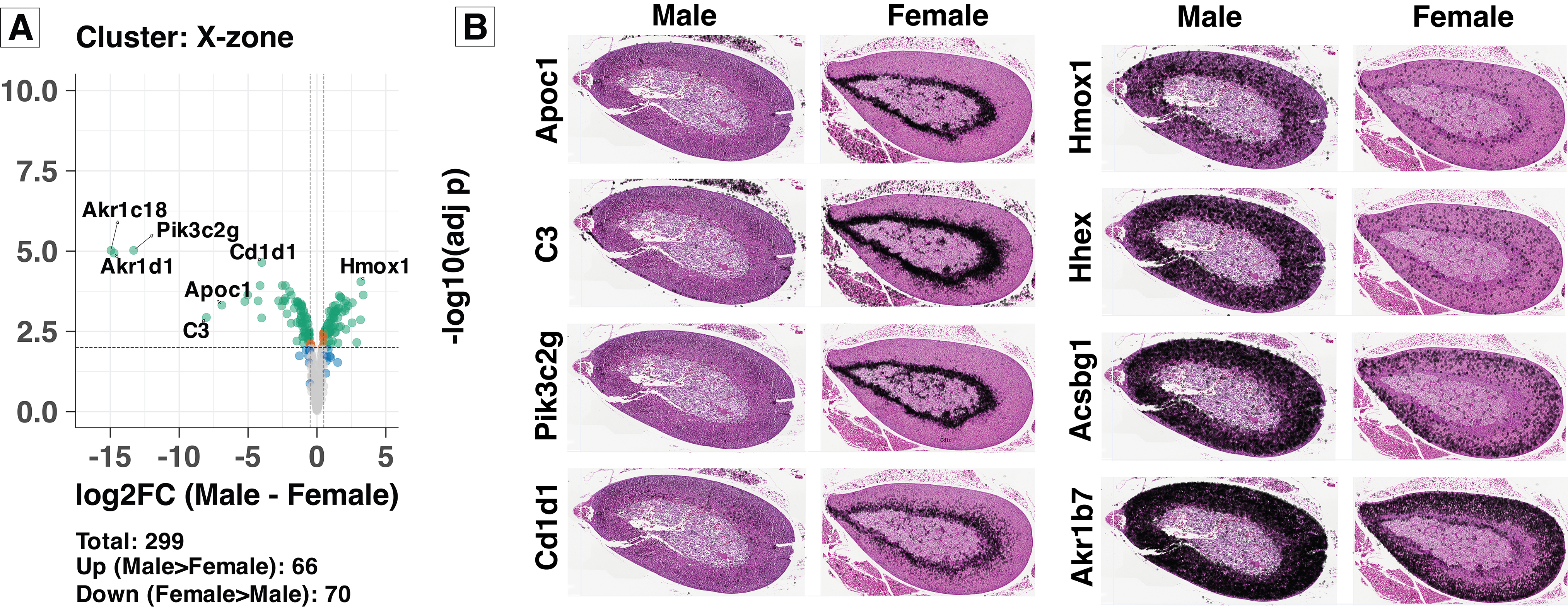
Przeprowadziliśmy kompleksową analizę gruczołu nadnerczy myszy z uwzględnieniem różnic płciowych, wykorzystując platformę Visium HD (10x Genomics). We conducted a comprehensive analysis of mouse adrenal gland including sex differences using the Visium HD platform (10x Genomics).
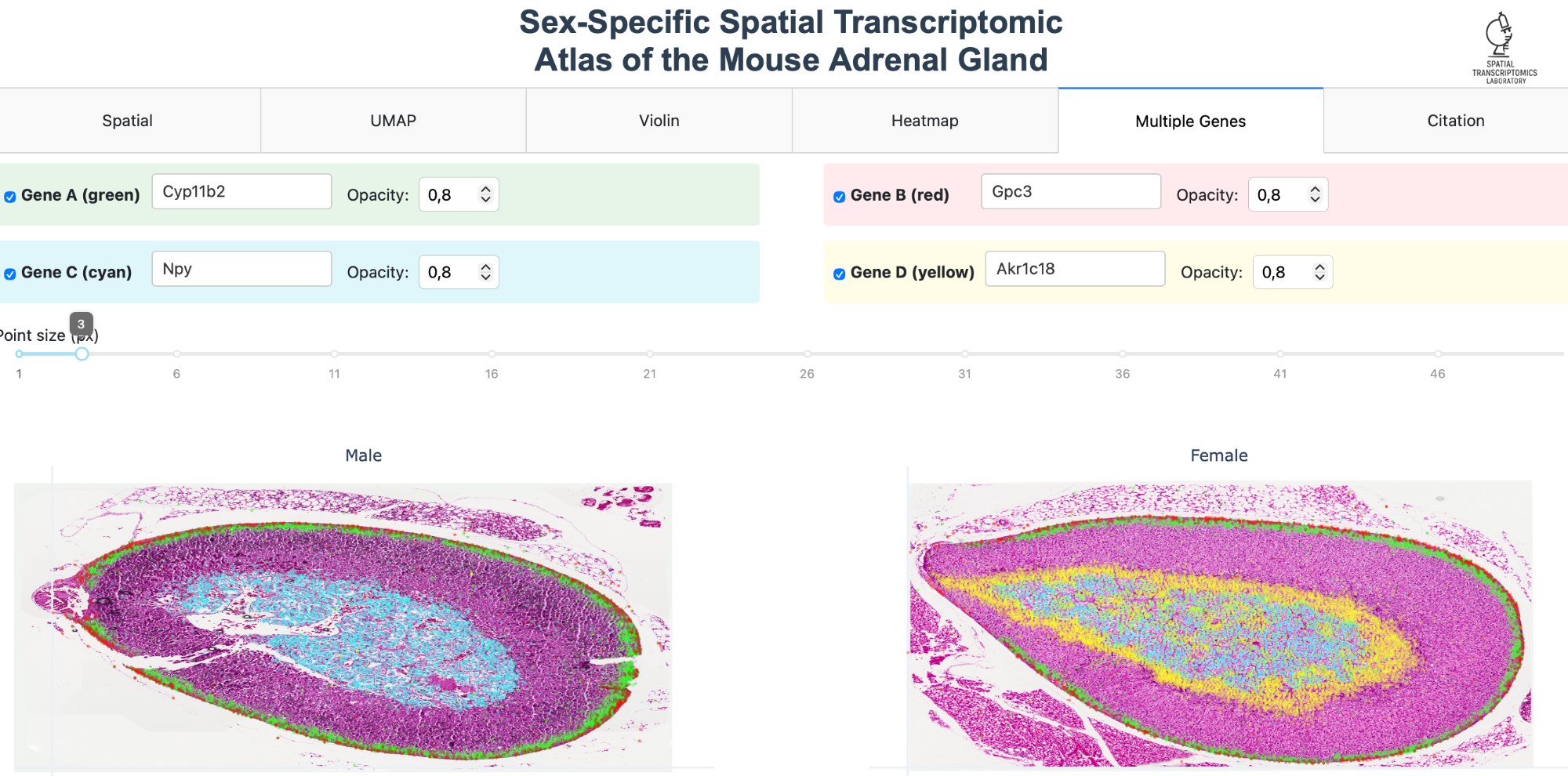
Eksploruj pełny atlas transkryptomiczny bezpośrednio w przeglądarce Explore the full transcriptomic atlas directly in your browser
Otwórz Atlas Open Atlas(tradycyjne Visium) (traditional Visium)
➜ Przejdź do atlasu ➜ Go to atlas➜ Przejdź do atlasu ➜ Go to atlas
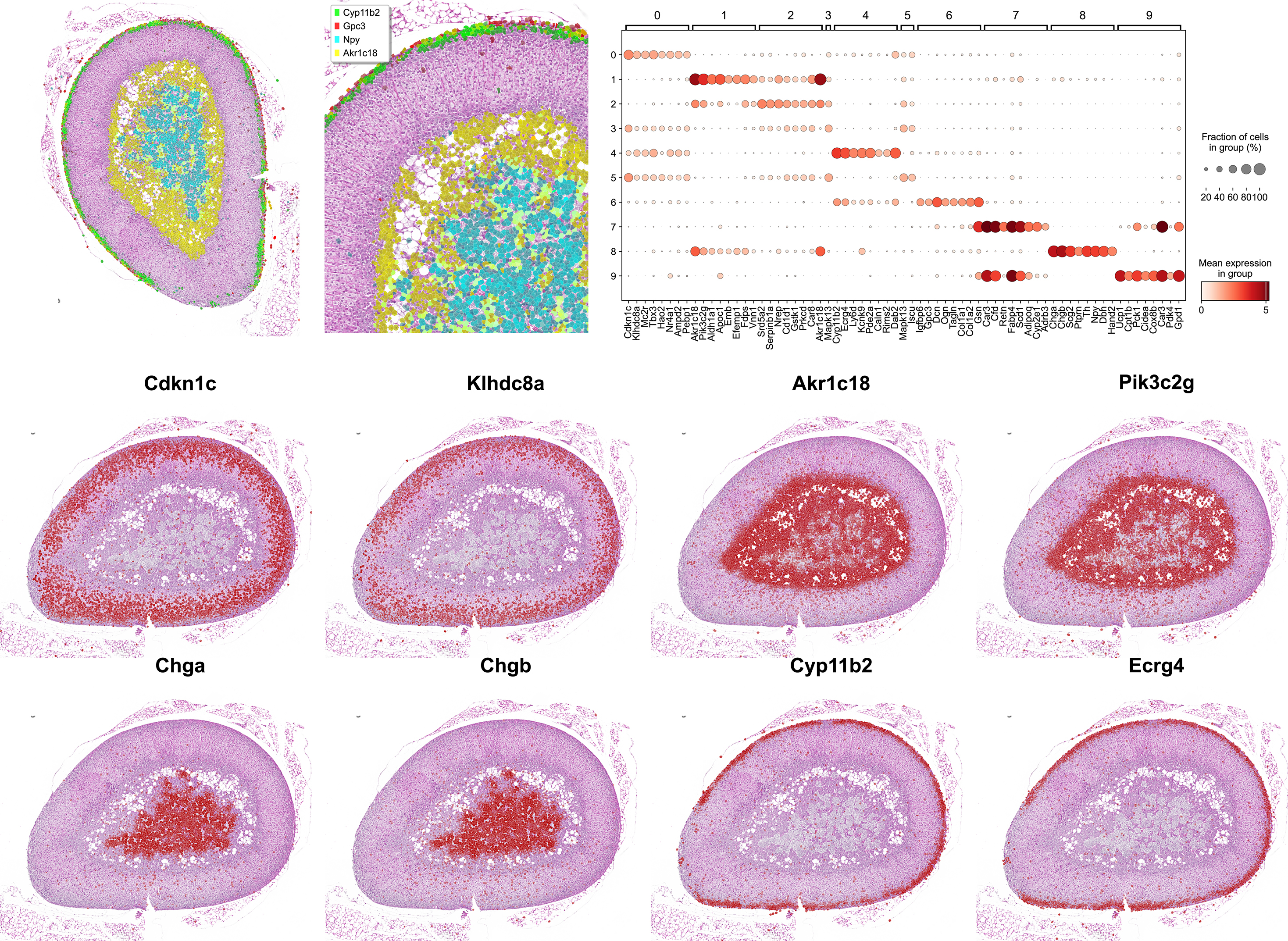
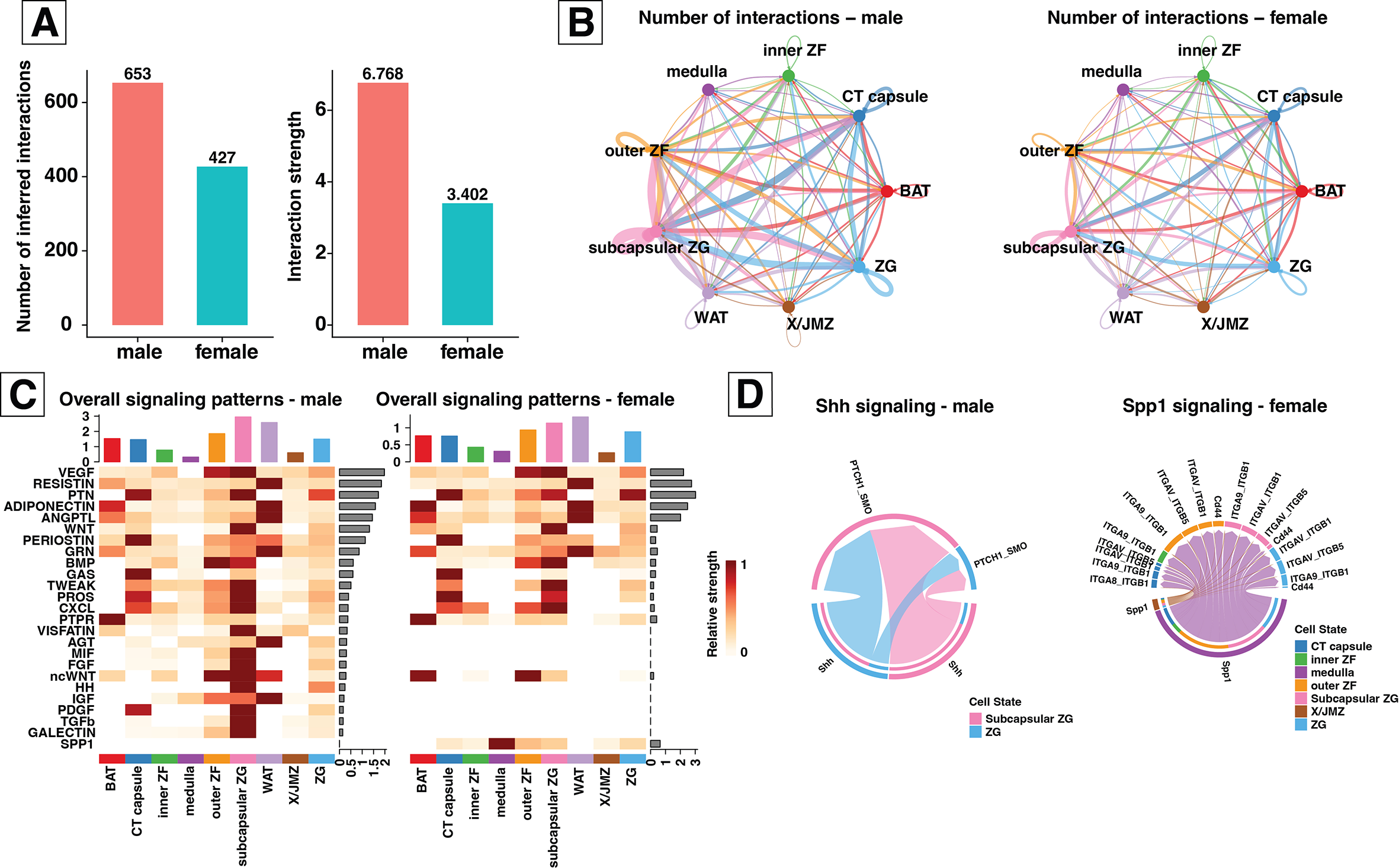
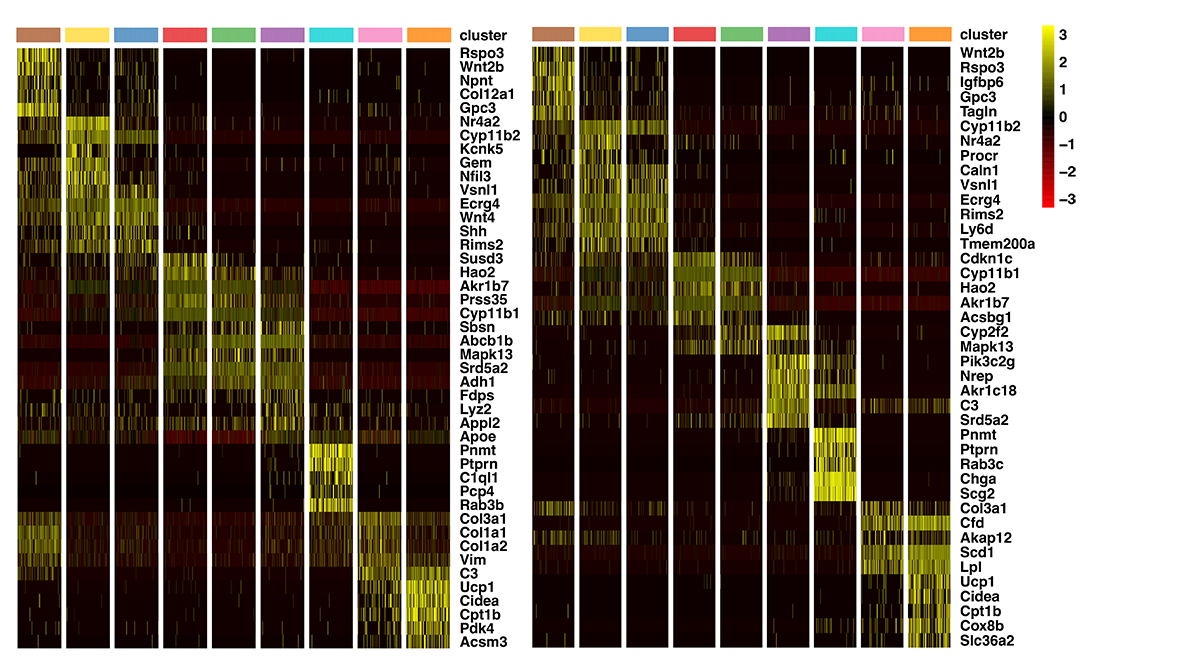




Transkryptomika przestrzenna znajduje szczególnie bogate zastosowanie w badaniach nad nowotworami, gdzie przestrzenna heterogenność odgrywa kluczową rolę. Spatial transcriptomics finds particularly rich applications in cancer research, where spatial heterogeneity plays a crucial role.
Oferujemy pełen zakres usług związanych z transkryptomiką przestrzenną, od projektowania eksperymentu po interaktywne atlasy. We offer a full range of services related to spatial transcriptomics, from experiment design to interactive atlases.
Skontaktuj się z nami, aby omówić Twój projekt Contact us to discuss your project
Zobacz Przykładowy Atlas View Sample Atlas